Les
documents ci-dessous montreront que la recherche pour créer COVID 19 a commencé
aux États-Unis en 2006 et a abouti à une bio-arme réussie en 2015, avec des
travaux effectués à l'Université de Caroline du Nord et à Harvard et au
laboratoire de la Food and Drug Administration en Arkansas. Leur travail était intitulé:
Un groupe de
coronavirus de chauves-souris en circulation ressemblant au SRAS montre un
potentiel d'émergence humaine
Ils ont
fait cela et bien plus encore, comme vous pourrez le lire ci-dessous.
Comme
Trump l'a dit, encore et encore, les Chinois étaient impliqués.
Laboratoire
clé des agents pathogènes spéciaux et de la biosécurité, Institut de virologie
de Wuhan, Académie chinoise des sciences, Wuhan ; la Chine a fourni le
virus de la chauve-souris de Wuhan qui a été utilisé dans l'étude américaine.
Leur nom (virus chinois) n'a été inclus que pour cette raison.
COVID 19
était un
projet d'armes biologiques de l'armée américaine visant à fabriquer
une pneumonie causant une maladie qu'il serait presque impossible de vacciner
chez des patients de plus de 40 ans.
La
preuve est là, faites simplement défiler vers le bas. L'étude a été dirigée par
l'Université de Caroline du Nord et financée par USAID / CIA. Ils ont choisi un
virus de chauve-souris chinois et ont également choisi d'inclure un centre
médical à Wuhan.
Maintenant,
nous savons pourquoi, un écran de fumée de condamnations pour un programme
auquel la Chine avait peu ou rien à voir, quelque chose de sataniquement
mauvais et purement américain.
En
novembre 2015, une étude a été publiée décrivant la capacité de produire le
virus dont nous traitons actuellement. Parmi les nombreux participants, il y
avait un laboratoire à Wuhan, en Chine. Il a été répertorié dès le début comme
l'une des dizaines d’équipes, principalement américaines, travaillant sur ce
projet.
Cependant,
un participant clé a été laissé de côté, l'USAID.
On soupçonne, profondément, que l'USAID est un front pour la recherche
américaine sur la bio-guerre telle que celle faite à Tbilissi, en Géorgie et
ailleurs, très documentée [1]. C'est
la citation qui ajoute l'USAID au groupe de financement de la recherche.
Changer l'historique
20 novembre 2015
Dans la version de cet article initialement publiée en ligne,
les auteurs ont omis de mentionner une source de financement, le financement
USAID-EPT-PREDICT d'EcoHealth Alliance, à Z.-L.S. L'erreur a été corrigée pour
les versions imprimée, PDF et HTML de cet article.
Nous
allons maintenant présenter l'article biaisé de Pravda et, ci-dessous, l'étude
réelle prouvant la capacité de produire COVID-19, prouvant, une fois pour
toutes, que ce n'est
pas un virus naturel.
Quant à
savoir qui a fait quoi, ce n'est pas notre travail, mais nous prouvons,
catégoriquement, que lorsqu'un laboratoire chinois est mentionné, il est un
acteur mineur dans un effort américain, comme indiqué de manière exhaustive
ci-dessous.
Cela
rend peut-être le laboratoire de Wuhan complice de la guerre biologique.
De même,
lorsque Forbes Magazine et d'autres ont déclaré qu'ils pouvaient prouver que
COVID-19 était fabriqué naturellement, et bien sûr, ils avaient le même accès
que nous, nous soupçonnons qu'ils
font partie d'un effort de désinformation lié à l'USAID et à la guerre
biologique.
La
suspicion n'est pas la preuve. La preuve est une preuve mais il y a
suffisamment de preuves évidentes. Nos remerciements aux professionnels américains
de la santé qui ont vendu leur âme à l'armée américaine et à la CIA et qui nous
ont amenés là où nous en sommes maintenant, une nation brisée en mille morceaux.
Pravda.Ru:
De tels documents sont apparus en 2015 sur le site Web de la revue scientifique
Natura en 2015. Ensuite, les auteurs ont affirmé qu'après l'avènement du
virus du SRAS (2002-2003) et du syndrome respiratoire du Moyen-Orient (MERS) [2], les scientifiques étaient au courant du
risque de transmission interspécifique qui conduirait à une épidémie parmi les
populations.
Expérience de
laboratoire réussie
L'équipe
de recherche a notamment étudié les chauves-souris, qui sont les plus grands
incubateurs de coronavirus. Néanmoins, les chauves-souris ne pouvaient pas transmettre
le coronavirus à l'homme car elles ne pouvaient pas interagir avec les cellules
humaines avec les récepteurs ACE2.
Le matériel a également déclaré que les chauves-souris en fer à
cheval portent une souche de coronavirus du SRAS qui peut être transmise à
l'homme. Il a été nommé virus
SHC014-CoV.
Pour mieux étudier ce virus, les scientifiques ont copié le
coronavirus et l'ont infecté avec des souris de laboratoire. Les résultats ont montré
que le virus est vraiment capable de se lier aux cellules humaines avec les
récepteurs ACE2 et de se multiplier dans les cellules du système respiratoire.
Dans le
travail de recherche, il est noté que le matériel, les échantillons et l'équipement de laboratoire
qui ont été utilisés dans la recherche ont été obtenus auprès de l'Institut de
recherche médicale de l'armée sur les maladies infectieuses. Cependant,
il n’est pas encore possible de dire avec certitude que le virus qui a été
testé sur des souris de laboratoire est le même que le coronavirus SARS-Cove-2.
Politique de l'OTAN
Cependant,
des choses intéressantes peuvent être trouvées dans des documents antérieurs. Par exemple:
• Le
rapport d'activité de l'Alliance 2019 indique qu'en 2019, la première place de
l'Alliance dans la recherche et le développement était occupée par le thème de
la protection radiochimique et biologique (29%), déplaçant le problème
apparemment le plus pressant de l'Europe - la lutte contre le terrorisme (devenue
en 4e priorité).
• Un an
plus tôt, en 2018, la situation était exactement l'inverse: le terrorisme,
comme il se doit, était en premier lieu (28%) et la protection radiochimique et
biologique en quatrième (13%).
Comme
l'écrit le mouchard de Bruxelles dans la chaîne des télégrammes, “étant donné l'absence de raisons visibles pour
un changement aussi marqué des intérêts scientifiques, il y a deux options et
les deux sont désagréables:
• ou
bien l'OTAN agite maintenant le cinquième point, falsifiant les données pour
montrer "et nous nous sommes toujours préparés aux virus, nous sommes
modernes",
• ou bien,
même en 2019 dans l'alliance, Dieu me pardonne, ils savaient d'où viendraient
les ennuis.
Oui, la
première option est beaucoup plus réelle, mais, voyez-vous, les faits sont
surprenants. "
Читайте больше на https://www.pravda.ru/world/1482450-COVID19/
Recherche originale de 2015 non révisée et complète
Un groupe de
coronavirus de chauves-souris en circulation ressemblant au SRAS montre un
potentiel d'émergence humaine
- Vineet D Menachery,
- Boyd L Yount Jr,
- Kari Debbink,
- Sudhakar Agnihothram,
- Lisa E Gralinski,
- Jessica A Plante,
- Rachel L Graham,
- Trevor Scobey,
- Xing-Yi Ge,
- Eric F Donaldson,
- Scott H Randell,
- Antonio
Lanzavecchia,
- Wayne A Marasco,
- Zhengli-Li Shi &
- Ralph S Baric
Medicine
volume 21, pages1508–1513 (2015) Citer cet article
• Accès
de 1,05 m
• 94
citations
• 6156
Altmetric
•
Détails métriques
• Un
rectificatif à cet article a été publié le 06 avril 2016
• Cet article a été mis à jour
Résumé
L'émergence
du coronavirus du syndrome respiratoire aigu sévère (SRAS-CoV) et du syndrome
respiratoire du Moyen-Orient (MERS) -CoV souligne la menace d'événements de
transmission inter-espèces conduisant à des épidémies chez l'homme. Nous
examinons ici le potentiel de maladie d'un virus de type SRAS, SHC014-CoV, qui
circule actuellement dans les populations de chauves-souris chinoises en fer à
cheval1. En utilisant le système de génétique inverse SARS-CoV2, nous avons
généré et caractérisé un virus chimérique exprimant le pic du coronavirus de
chauve-souris SHC014 dans un squelette SARS-CoV adapté à la souris. Les
résultats indiquent que les virus du groupe 2b codant pour le pic SHC014 dans
un squelette de type sauvage peuvent utiliser efficacement plusieurs
orthologues de l'enzyme de conversion de l'angiotensine humaine II (ACE2), se
répliquer efficacement dans les cellules des voies respiratoires humaines
primaires et atteindre des titres in vitro équivalents à une épidémie souches
de SARS-CoV. De plus, des expériences in vivo démontrent la réplication du
virus chimérique dans le poumon de souris avec une pathogenèse notable.
L'évaluation des modalités immunothérapeutiques et prophylactiques basées sur
le SRAS a révélé une faible efficacité; les approches à base d'anticorps
monoclonaux et de vaccins n'ont pas réussi à neutraliser et à protéger contre
l'infection par les CoV en utilisant la nouvelle protéine de pointe. Sur la
base de ces résultats, nous avons dérivé synthétiquement un virus recombinant
SHC014 infectieux de pleine longueur et démontrons une réplication virale
robuste à la fois in vitro et in vivo. Nos travaux suggèrent un risque
potentiel de réémergence du SRAS-CoV à partir de virus circulant actuellement
dans les populations de chauves-souris.
Partie Principale
L'émergence
du SRAS-CoV a inauguré une nouvelle ère dans la transmission interespèces de
maladies respiratoires graves, la mondialisation entraînant une propagation rapide
dans le monde et un impact économique massif3,4. Depuis lors, plusieurs souches
- dont les souches de grippe A H5N1, H1N1 et H7N9 et MERS-CoV - ont émergé des
populations animales, causant des maladies, une mortalité et des difficultés
économiques considérables pour les régions touchées5. Bien que des mesures de
santé publique aient pu arrêter l'épidémie de SRAS-CoV4, de récentes études
métagénomiques ont identifié des séquences de virus de type SRAS étroitement
apparentés circulant dans les populations de chauves-souris chinoises qui
pourraient constituer une menace future1,6. Cependant, les données de séquence
à elles seules fournissent un minimum d'informations pour identifier et
préparer les futurs virus prépandémiques. Par conséquent, pour examiner le
potentiel d'émergence (c'est-à-dire le potentiel d'infecter les humains) des
CoV de chauve-souris en circulation, nous avons construit un virus chimérique
codant pour une nouvelle protéine de pointe de CoV zoonotique - à partir de la
séquence RsSHC014-CoV qui a été isolée des chauves-souris chinoises en fer à
cheval1 - dans le cadre du squelette SARS-CoV adapté aux souris. Le virus
hybride nous a permis d'évaluer la capacité de la nouvelle protéine de pointe à
provoquer une maladie indépendamment des autres mutations adaptatives
nécessaires dans son squelette naturel. En utilisant cette approche, nous avons
caractérisé l'infection au CoV médiée par la protéine de pointe SHC014 dans les
cellules des voies aériennes humaines primaires et in vivo, et testé
l'efficacité des thérapies immunitaires disponibles contre SHC014-CoV.
Ensemble, la stratégie traduit les données de métagénomique pour aider à
prévoir et à préparer les futurs virus émergents.
Les
séquences de SHC014 et du RsWIV1-CoV apparenté montrent que ces CoV sont les
plus proches parents des souches épidémiques du SRAS-CoV (Fig. 1a, b);
cependant, il existe des différences importantes dans les 14 résidus qui se
lient à l'ACE2 humain, le récepteur du SRAS-CoV, y compris les cinq qui sont
critiques pour la gamme d'hôtes: Y442, L472, N479, T487 et Y491 (réf.7). Dans
WIV1, trois de ces résidus diffèrent de la souche épidémique SARS-CoV Urbani,
mais on ne s'attendait pas à ce qu'ils altèrent la liaison à ACE2 (Fig. 1a, b et Tableau 1). Ce fait
est confirmé à la fois par des expériences de pseudotypage qui ont mesuré la
capacité des lentivirus codant pour des protéines de pointe WIV1 à pénétrer
dans des cellules exprimant l'ACE2 humaine (Fig.1 supplémentaire) et par des
tests de réplication in vitro de WIV1-CoV (réf.1). En revanche, 7 des 14
résidus d'interaction ACE2 dans SHC014 sont différents de ceux dans SARS-CoV, y
compris les cinq résidus critiques pour la gamme d'hôtes (figure supplémentaire
1c et tableau supplémentaire 1). Ces changements, couplés à l'échec des
lentivirus pseudotypés exprimant la pointe SHC014 à pénétrer dans les cellules
(figure supplémentaire 1d), suggèrent que la pointe SHC014 est incapable de se
lier à l'ACE2 humain. Cependant, des changements similaires dans les souches
associées du SRAS-CoV avaient été signalés pour permettre la liaison de
l'ACE27,8, suggérant que des tests fonctionnels supplémentaires étaient
nécessaires pour la vérification. Par conséquent, nous avons synthétisé la
pointe SHC014 dans le contexte du squelette SARS-CoV adapté à la réplication
(adapté à la souris) (nous désignerons ci-après le CoV chimérique SHC014-MA15)
afin de maximiser l'opportunité d'études de pathogenèse et de vaccins chez la
souris (Fig supplémentaire . 2a). Malgré les prédictions à la fois de la
modélisation basée sur la structure et des expériences de pseudotypage,
SHC014-MA15 était viable et répliqué à des titres élevés dans les cellules Vero
(figure supplémentaire 2b). De façon similaire au SRAS, SHC014-MA15 nécessitait
également une molécule ACE2 fonctionnelle pour l'entrée et pouvait utiliser des
orthologues ACE2 humains, civettes et chauves-souris (figure supplémentaire 2c,
d). Pour tester la capacité de la pointe SHC014 à médier l'infection des voies
respiratoires humaines, nous avons examiné la sensibilité de la lignée
cellulaire des voies respiratoires épithéliales humaines Calu-3 2B4 (réf. 9) à
l'infection et trouvé une réplication SHC014-MA15 robuste, comparable à celle
de SARS-CoV Urbani (figure 1c). Pour étendre ces résultats, les cultures
primaires d'épithélium des voies respiratoires humaines (AOH) ont été infectées
et ont montré une réplication robuste des deux virus (Fig. 1d). Ensemble, les
données confirment la capacité des virus avec la pointe SHC014 à infecter les
cellules des voies respiratoires humaines et soulignent la menace potentielle
de transmission inter-espèces de SHC014-CoV.
 Figure 1: Les virus de
type SRAS se répliquent dans les cellules des voies respiratoires humaines et
produisent une pathogenèse in vivo.
Figure 1: Les virus de
type SRAS se répliquent dans les cellules des voies respiratoires humaines et
produisent une pathogenèse in vivo.
(a) Les
séquences génomiques complètes de CoV représentatifs ont été alignées et
cartographiées phylogénétiquement comme décrit dans les Méthodes en ligne. La
barre d'échelle représente les substitutions de nucléotides, seul le support
bootstrap supérieur à 70% étant marqué. L'arbre montre les CoV divisés en trois
groupes phylogénétiques distincts, définis comme α-CoV, β-CoV et γ-CoV. Les grappes de sous-groupes classiques
sont marquées comme 2a, 2b, 2c et 2d pour les β-CoV et comme 1a et 1b pour
les α-CoV. (b) Les séquences d'acides aminés des domaines S1 des
pointes des β-CoV représentatifs du groupe 2b, y compris le SARS-CoV, ont été
alignées et cartographiées phylogénétiquement. La barre d'échelle représente
les substitutions d'acides aminés. (c, d) Réplication virale de SARS-CoV
Urbani (noir) et SHC014-MA15 (vert) après infection de cellules Calu-3 2B4 (c)
ou cultures de cellules HAE à interface air-liquide primaire bien différenciées
(d) à une multiplicité d'infection (MOI) de 0,01 pour les deux types de
cellules. Des échantillons ont été prélevés à des moments individuels avec des
répliques biologiques (n = 3) pour les expériences Calu-3 et HAE. (e, f) Perte
de poids (n = 9 pour SARS-CoV MA15; n = 16 pour SHC014-MA15) (e) et réplication
virale dans les poumons (n = 3 pour SARS-CoV MA15; n = 4 pour SHC014- MA15)
(f) de souris BALB / c âgées de 10 semaines infectées par 1 × 104 pfu de
SARS-CoV MA15 adapté aux souris (noir) ou SHC014-MA15 (vert) par voie intranasale
(i.n.). (g, h) Des images représentatives de coupes pulmonaires colorées pour
l'antigène SARS-CoV N de souris infectées par SARS-CoV MA15 (n = 3 souris) (g)
ou SHC014-MA15 (n = 4 souris) (h) sont montrées. Pour chaque graphique, la
valeur centrale représente la moyenne du groupe et les barres d'erreur définissent
la valeur s.e.m. Barres d'échelle, 1 mm.
Pour
évaluer le rôle du pic SHC014 dans la médiation de l'infection in vivo, nous
avons infecté des souris BALB / c âgées de 10 semaines avec 104 unités
formatrices de plaque (pfu) de SARS-MA15 ou SHC014-MA15 (Fig. 1e – h) . Les
animaux infectés par le SRAS-MA15 ont connu une perte de poids rapide et une
létalité 4 jours après l'infection (d.p.i.); en revanche, l'infection par
SHC014-MA15 a produit une perte de poids substantielle (10%) mais aucune
létalité chez la souris (figure 1e). L'examen de la réplication virale a révélé
des titres viraux presque équivalents dans les poumons de souris infectées par
le SRAS-MA15 ou SHC014-MA15 (figure 1f). Alors que les poumons des souris
infectées par le SRAS-MA15 présentaient une coloration robuste à la fois dans
les bronchioles terminales et dans le parenchyme pulmonaire 2 d.p.i. (Fig. 1g),
celles des souris infectées par SHC014-MA15 ont montré une diminution de la
coloration de l'antigène des voies respiratoires (Fig. 1h); en revanche, aucun
déficit dans la coloration de l'antigène n'a été observé dans le parenchyme ou
dans le score histologique global, suggérant une infection différentielle du
tissu pulmonaire pour SHC014-MA15 (tableau supplémentaire 2). Nous avons
ensuite analysé l'infection chez des animaux âgés (12 mois) plus sensibles. Les
animaux infectés par le SRAS-MA15 ont rapidement perdu du poids et succombé à
l'infection (figure supplémentaire 3a, b). L'infection à SHC014-MA15 a induit
une perte de poids robuste et soutenue, mais avait une létalité minimale. Les
tendances de l'histologie et des profils de coloration de l'antigène que nous avons
observées chez les jeunes souris ont été conservées chez les animaux plus âgés
(tableau supplémentaire 3). Nous avons exclu la possibilité que SHC014-MA15
soit médiatrice de l'infection par un récepteur alternatif sur la base
d'expériences utilisant des souris Ace2 - / -, qui n'ont pas montré de perte de
poids ou de coloration d'antigène après une infection par SHC014-MA15 (Fig. 4a,
b et Supplémentaire supplémentaires). Tableau 2). Ensemble, les données
indiquent que les virus avec le pic SHC014 sont capables d'induire une perte de
poids chez la souris dans le contexte d'un squelette CoV virulent.
Compte
tenu de l'efficacité préclinique des thérapies par anticorps monoclonaux contre
Ebola, telles que ZMApp10, nous avons ensuite cherché à déterminer l'efficacité
des anticorps monoclonaux contre le SRAS-CoV contre l'infection par
SHC014-MA15.
Quatre
anticorps monoclonaux humains largement neutralisants ciblant la protéine de
pointe du SRAS-CoV avaient déjà été signalés et sont des réactifs probables
pour l'immunothérapie11,12,13. Nous avons examiné l'effet de ces anticorps sur
la réplication virale (exprimé en pourcentage d'inhibition de la réplication
virale) et avons constaté que, alors que le SARS-CoV Urbani de type sauvage
était fortement neutralisé par les quatre anticorps à des concentrations
d'anticorps relativement faibles (Fig.2a-d), la neutralisation variait pour
SHC014-MA15. Fm6, un anticorps généré par l'affichage des phages et les mutants
d'échappement 11,12, n'a atteint que des niveaux de fond d'inhibition de la
réplication SHC014-MA15 (figure 2a). De même, les anticorps 230.15 et 227.14,
qui étaient dérivés de cellules mémoire B de patients infectés par le
SRAS-CoV13, n'ont pas non plus bloqué la réplication de SHC014-MA15 (Fig. 2b,
c). Pour les trois anticorps, les différences entre les séquences d'acides
aminés des pics SARS et SHC014 correspondaient à des changements de résidus
directs ou adjacents trouvés dans les mutants d'échappement SARS-CoV (fm6
N479R; 230.15 L443V; 227.14 K390Q / E), ce qui explique probablement l'absence
des anticorps 'activité neutralisante contre SHC014. Enfin, l'anticorps
monoclonal 109.8 a pu atteindre une neutralisation à 50% de SHC014-MA15, mais
uniquement à des concentrations élevées (10 μg / ml) (Fig. 2d). Ensemble, les résultats
démontrent que les anticorps neutralisants à grande échelle contre le SRAS-CoV
ne peuvent avoir qu'une efficacité marginale contre les souches émergentes de
CoV de type SRAS telles que SHC014.
Figure 2: Les
anticorps monoclonaux contre le SRAS-CoV ont une efficacité marginale contre
les CoV de type SRAS.
(a–d) Essais de neutralisation évaluant
l'efficacité (mesurée en tant que réduction du nombre de plaques) d'un panel
d'anticorps monoclonaux, qui ont tous été initialement générés contre le
SRAS-CoV épidémique, contre l'infection des cellules Vero par le SARS-CoV
Urbani (noir) ou SHC014-MA15 (vert). Les anticorps testés étaient fm6 (n = 3
pour Urbani; n = 5 pour SHC014-MA15) 11,12 (a), 230,15 (n = 3
pour Urbani; n = 2 pour SHC014-MA15) (b), 227,15 (n = 3 pour Urbani; n =
5 pour SHC014-MA15) (c) et 109,8 (n = 3 pour Urbani; n = 2 pour SHC014-MA15) 13
(d). Chaque point de données représente la moyenne du groupe et les
barres d'erreur définissent la valeur s.e.m. Notez que les barres d'erreur dans
les cellules Vero infectées par le SRAS-CoV Urbani en b, c sont
superposées par les symboles et ne sont pas visibles.
Pour
évaluer l'efficacité des vaccins existants contre l'infection par SHC014-MA15,
nous avons vacciné des souris âgées avec du SARS-CoV (DIV) entier double-inactivé.
Des travaux antérieurs ont montré que DIV pouvait neutraliser et protéger les
jeunes souris contre la provocation par un virus homologue14; cependant, le
vaccin n'a pas réussi à protéger les animaux âgés chez lesquels une pathologie
immunitaire augmentée a également été observée, indiquant la possibilité que
les animaux soient blessés en raison de la vaccination15. Ici, nous avons
constaté que DIV n'a pas fourni de protection contre la provocation avec
SHC014-MA15 en ce qui concerne la perte de poids ou le titre viral (Fig.
Supplémentaire 5a, b). Conformément à un rapport précédent avec d'autres CoVs15 hétérologues du groupe 2b,
le sérum de souris âgées vaccinées par DIV n'a pas non plus neutralisé
SHC014-MA15 (figure supplémentaire 5c). Notamment, la vaccination DIV a
entraîné une pathologie immunitaire robuste (tableau supplémentaire 4) et une
éosinophilie (figure supplémentaire 5d – f). Ensemble, ces résultats confirment
que le vaccin DIV ne protégerait pas contre l'infection par SHC014 et pourrait
éventuellement augmenter la maladie dans le groupe vacciné âgé.
Contrairement
à la vaccination des souris avec DIV, l'utilisation de SHC014-MA15 comme vaccin
vivant atténué a montré une protection croisée potentielle contre la
provocation par le SRAS-CoV, mais les résultats comportent d'importantes
réserves. Nous avons infecté de jeunes souris avec 104 p.f.u. de SHC014-MA15 et
les a observés pendant 28 jours. Nous avons ensuite provoqué les souris avec
SARS-MA15 au jour 29 (Fig. Supplémentaire 6a). L'infection antérieure des
souris par la dose élevée de SHC014-MA15 a conféré une protection contre la
provocation par une dose létale de SRAS-MA15, bien qu'il n'y ait eu qu'une
réponse de neutralisation minimale du SRAS-CoV des antisérums déclenchée 28
jours après l'infection par SHC014-MA15 ( Fig.6b supplémentaire, 1: 200). En
l'absence de rappel d'antigène secondaire, 28 d.p.i. représente le pic attendu
des titres d'anticorps et implique que la protection contre le SRAS-CoV
diminuera avec le temps16,17. Des résultats similaires
montrant une protection contre la provocation avec une dose létale de SARS-CoV
ont été observés chez des souris BALB / c âgées en ce qui concerne la perte de
poids et la réplication virale (figure supplémentaire 6c, d). Cependant, la
dose d'infection SHC014-MA15 de 104 p.f.u. induit> 10% de perte de poids et
de létalité chez certains animaux âgés (Fig. 1 et Fig. 3 supplémentaire). Nous
avons constaté que la vaccination avec une dose plus faible de SHC014-MA15 (100
p.f.u.), n'induisait pas de perte de poids, mais elle ne protégeait pas non
plus les animaux âgés d'une provocation par la dose létale du SRAS-MA15 (figure
supplémentaire 6e, f). Ensemble, les données suggèrent que la provocation par
SHC014-MA15 peut conférer une protection croisée contre le SRAS-CoV par le
biais d'épitopes conservés, mais la dose requise induit la pathogenèse et
empêche l'utilisation comme vaccin atténué.
Après
avoir établi que la pointe SHC014 a la capacité de médier l'infection des
cellules humaines et de provoquer des maladies chez la souris, nous avons ensuite
synthétisé un clone infectieux SHC014-CoV de pleine longueur basé sur
l'approche utilisée pour le SRAS-CoV (Fig.3a) 2. La réplication dans les
cellules Vero n'a révélé aucun déficit pour SHC014-CoV par rapport à celui pour
SARS-CoV (Fig. 3b); cependant, SHC014-CoV était significativement (P <0 24="" 3c="" 3d="" 48="" a="" adaptation="" apr="" att="" au="" aucune="" celle="" cellules="" cessaire="" chez="" compl="" cultures="" d="" dans="" de="" du="" duite="" e="" ensemble="" est="" et="" fois="" h="" humaines="" ig.="" in="" infection="" l="" la="" les="" longueur="" mais="" mentaire="" mique="" montr="" n="" nu="" par="" perte="" pid="" pleine="" plication="" poids="" poumons="" pour="" primaires="" qu="" que="" quivalente="" r="" rapport="" rent="" respiratoires="" s="" sa="" sars-cov="" shc014-cov="" significative="" soit="" souris.="" souris="" span="" sugg="" sultats="" suppl="" tablissent="" te="" une="" urbani="" viabilit="" virale="" vivo="">
Figure 3: SHC014-CoV
pleine longueur se réplique dans les voies respiratoires humaines mais n'a pas
la virulence du SRV-CoV épidémique.
(a)
Schéma du clone moléculaire SHC014-CoV, qui a été synthétisé sous la forme de
six ADNc contigus (désignés SHC014A, SHC014B, SHC014C, SHC014D, SHC014E et
SHC014F) flanqués de sites BglI uniques qui ont permis l'assemblage dirigé de
l'ADNc pleine longueur exprimant cadres de lecture ouverts (pour 1a, 1b, spike,
3, enveloppe, matrice, 6–8 et nucléocapside). Les nucléotides soulignés
représentent les séquences en surplomb formées après le clivage de l'enzyme de
restriction. (b, c) Réplication virale de SARS-CoV Urbani (noir) ou
SHC014-CoV (vert) après infection de cellules Vero (b) ou cultures de
cellules HAE à interface air-liquide primaire bien différenciées (c) à
un MOI de 0,01. Des échantillons ont été prélevés à des moments individuels
avec des répliques biologiques (n = 3) pour chaque groupe. Les données
représentent une expérience pour les cellules Vero et HAE. (d, e) Perte
de poids (n = 3 pour SARS-CoV MA15, n = 7 pour SHC014-CoV; n = 6 pour SARS-Urbani)
(d) et réplication virale dans les poumons (n = 3 pour
SARS-Urbani et SHC014-CoV) (e) de souris BALB / c âgées de 10 semaines infectées
par 1 × 105 pfu de SARS-CoV MA15 (gris),
SHC014-CoV (vert) ou SARS-CoV Urbani (noir) via le i.n. route. Chaque point de
données représente la moyenne du groupe et des barres d'erreur définissent la
valeur s.e.m. ** P <0 bilat="" de="" en="" et="" individuels.="" le="" p="" points="" ral="" span="" student="" t="" temporels="" test="" utilisant="">
Pendant
l'épidémie de SRAS-CoV, des liens ont été rapidement établis entre les civettes
de palmier et les souches de CoV détectées chez l'homme4. S'appuyant sur cette
découverte, le paradigme d'émergence commun soutient que le SRAS-CoV épidémique
est originaire d'un virus de chauve-souris, a sauté aux civettes et a incorporé
des changements dans le domaine de liaison aux récepteurs (RBD) pour améliorer
la liaison à la civette Ace2 (réf.
18). Une exposition subséquente
à des personnes sur les marchés d'animaux vivants a permis à l'infection
humaine de la souche civette, qui, à son tour, s'est adaptée pour devenir la
souche épidémique (Fig. 4a). Cependant, l'analyse
phylogénétique suggère que les premières souches humaines du SRAS semblent plus
étroitement liées aux souches de chauves-souris qu'aux souches de civette18. Par conséquent, un deuxième
paradigme soutient que la transmission directe chauve-souris humaine a
déclenché l'émergence du SRAS-CoV et que les civettes de palmier ont servi
d'hôte secondaire et de réservoir pour une infection continue (Fig. 4b)19. Pour les deux paradigmes,
l'adaptation des pointes chez un hôte secondaire est considérée comme une
nécessité, la plupart des mutations devant se produire dans le RBD, facilitant
ainsi une infection améliorée. Les deux théories impliquent que les pools de
CoV des chauves-souris sont limités et que les mutations de la gamme d'hôtes sont
à la fois aléatoires et rares, ce qui réduit la probabilité de futurs
événements d'émergence chez l'homme.
Figure 4: Paradigmes
d'émergence pour les coronavirus.
Les
souches de coronavirus sont maintenues dans des pools de quasi-espèces circulant
dans les populations de chauves-souris. (a, b) Les théories
traditionnelles sur l'émergence du SRAS-CoV supposent que les mutants de la
gamme d'hôtes (cercle rouge) représentent des occurrences aléatoires et rares
qui permettent l'infection d'hôtes alternatifs. Le paradigme de l'hôte
secondaire (a) soutient qu'un hôte non humain est infecté par un virus
progéniteur de chauve-souris et, par l'adaptation, facilite la transmission à
l'homme; la réplication ultérieure chez l'homme conduit à la souche virale
épidémique. Le paradigme direct (b) suggère que la transmission se
produit entre les chauves-souris et les humains sans avoir besoin d'un hôte
intermédiaire; la sélection se produit alors dans la population humaine avec
des virus étroitement apparentés se répliquant dans un hôte secondaire,
permettant une persistance et une adaptation virales continues dans les deux. (c)
Les données des virus chimériques de type SRAS font valoir que les pools de
quasi-espèces maintiennent plusieurs virus capables d'infecter les cellules
humaines sans avoir besoin de mutations (cercles rouges). Bien que des
adaptations chez des hôtes secondaires ou humains puissent être nécessaires
pour l'émergence d'une épidémie, si des virus contenant des pointes SHC014 sont
recombinés avec des squelettes CoV virulents (cercles avec des contours verts),
alors la maladie épidémique peut être le résultat chez l'homme. Les données
existantes prennent en charge les éléments des trois paradigmes.
Bien que
notre étude n'invalide pas les autres voies d'émergence, elle plaide pour un
troisième paradigme dans lequel les pools de CoV de chauves-souris en
circulation maintiennent des protéines de pointe `` en équilibre '' qui sont
capables d'infecter les humains sans mutation ni adaptation (Fig. 4c). Cette hypothèse est
illustrée par la capacité d'un virus chimérique contenant la pointe SHC014 dans
un squelette SARS-CoV à provoquer une infection robuste dans les cultures des
voies respiratoires humaines et chez les souris sans adaptation RBD. Couplés à
l'observation de squelettes CoV pathogènes précédemment identifiés3,20, nos
résultats suggèrent que les matériaux de départ requis pour les souches
émergentes de type SRAS circulent actuellement dans des réservoirs d'animaux.
Notamment, bien que SHC014-CoV de pleine longueur nécessite probablement une
adaptation supplémentaire du squelette pour médier la maladie humaine, les
événements de recombinaison à haute fréquence documentés dans les familles de
CoV soulignent la possibilité d'émergence future et la nécessité d'une
préparation supplémentaire.
À ce
jour, les dépistages génomiques des populations animales ont été principalement
utilisés pour identifier de nouveaux virus dans les foyers21. L'approche ici étend ces
ensembles de données pour examiner les questions d'émergence virale et
d'efficacité thérapeutique. Nous considérons les virus avec le pic SHC014 comme
une menace potentielle en raison de leur capacité à se répliquer dans les
cultures des voies respiratoires humaines primaires, le meilleur modèle
disponible pour les maladies humaines. De plus, la pathogenèse observée chez la
souris indique une capacité des virus contenant SHC014 à provoquer des maladies
dans les modèles mammifères, sans adaptation RBD. Notamment, le tropisme
différentiel dans le poumon par rapport à celui avec le SRAS-MA15 et
l'atténuation du SHC014-CoV de pleine longueur dans les cultures d'AOH par
rapport au SARS-CoV Urbani suggèrent que des facteurs au-delà de la liaison
ACE2, y compris la processivité des pointes, la biodisponibilité des récepteurs
ou l'antagonisme des réponses immunitaires de l'hôte - peut contribuer à
l'émergence. Cependant, des tests supplémentaires sur des primates non humains
sont nécessaires pour traduire ces résultats en potentiel pathogène chez
l'homme. Surtout, l'échec des thérapies disponibles définit un besoin critique
pour une étude plus approfondie et pour le développement de traitements. Grâce
à ces connaissances, des programmes de surveillance, des réactifs de diagnostic
et des traitements efficaces peuvent être produits qui protègent contre
l'émergence de CoV spécifiques au groupe 2b, tels que SHC014, et ceux-ci
peuvent être appliqués à d'autres branches de CoV qui maintiennent des pools
hétérogènes similaires.
En plus
d'offrir une préparation contre les futurs virus émergents, cette approche doit
être envisagée dans le contexte de la pause imposée par le gouvernement
américain sur les études de gain de fonction (GOF)22. Sur la base des modèles
d'émergence précédents (Fig. 4a,b), la création de virus
chimériques tels que SHC014-MA15 ne devrait pas augmenter la pathogénicité.
Bien que SHC014-MA15 soit atténué par rapport à son SARS-CoV adapté à la souris
parentale, des études similaires examinant la pathogénicité des CoV avec la
pointe Urbani de type sauvage dans le squelette MA15 n'ont montré aucune perte
de poids chez la souris et une réplication virale réduite23. Ainsi, par rapport au pic
Urbani-MA15 CoV, SHC014-MA15 montre un gain de pathogenèse (Fig. 1). Sur la base de ces
résultats, les comités d'examen scientifique peuvent juger les études
similaires construisant des virus chimériques basés sur des souches en
circulation trop risquées, car une pathogénicité accrue dans les modèles
mammifères ne peut être exclue. Couplé à des restrictions sur les souches
adaptées à la souris et au développement d'anticorps monoclonaux utilisant des
mutants d'échappement, la recherche sur l'émergence de CoV et l'efficacité
thérapeutique pourrait être sérieusement limitée à l'avenir. Ensemble, ces
données et restrictions représentent un carrefour des préoccupations de
recherche du GOF; le potentiel de préparation et d'atténuation des flambées
futures doit être mis en balance avec le risque de création d'agents pathogènes
plus dangereux. Lors de l'élaboration de politiques à l'avenir, il est
important de tenir compte de la valeur des données générées par ces études et
de savoir si ces types d'études sur le virus chimérique justifient une enquête
plus approfondie par rapport aux risques inhérents impliqués.
Dans
l'ensemble, notre approche a utilisé des données de métagénomique pour
identifier une menace potentielle posée par la chauve-souris circulante de type
SRV CoC SHC014. En raison de la capacité des virus chimériques SHC014 à se
répliquer dans les cultures des voies respiratoires humaines, à provoquer une
pathogenèse in vivo et à échapper aux thérapies actuelles, il existe un besoin
à la fois de surveillance et de thérapies améliorées contre les virus de type
SRAS en circulation. Notre approche permet également de débloquer l'utilisation
des données métagénomiques pour prédire l'émergence virale et d'appliquer ces
connaissances dans la préparation du traitement des futures infections virales
émergentes.
Les méthodes
Virus, cellules,
infection in vitro et tests sur plaque.
Le
SARS-CoV de type sauvage (Urbani), le SARS-CoV adapté aux souris (MA15) et les
CoV chimériques de type SARS ont été cultivés sur des cellules Vero E6
(obtenues auprès de l'Institut de recherche médicale des États-Unis sur les
maladies infectieuses), cultivées dans le Eagle's modifié de Dulbecco. moyen
(DMEM) (Gibco, CA) et 5% de sérum de clone fœtal (FCS) (Hyclone, South Logan,
UT) avec un antibiotique / antimycotique (Gibco, Carlsbad, CA). Des cellules
DBT (laboratoire barique, source inconnue) exprimant des orthologues ACE2 ont
été précédemment décrites pour l'homme et la civette; la séquence bat Ace2 était
basée sur celle de Rhinolophus leschenaulti, et les cellules DBT
exprimant la batte Ace2 ont été établies comme décrit précédemment8. Les expériences de
pseudotypage étaient similaires à celles utilisant un pseudovirus basé sur le
VIH, préparées comme décrit précédemment10, et examinées sur des
cellules HeLa (Wuhan Institute of Virology) qui exprimaient des orthologues
ACE2. Les cellules HeLa ont été cultivées dans un milieu essentiel minimal
(MEM) (Gibco, CA) additionné de 10% de FCS (Gibco, CA) comme décrit
précédemment24. Les courbes de croissance
dans Vero E6, DBT, Calu-3 2B4 et les cellules épithéliales des voies aériennes
humaines primaires ont été réalisées comme décrit précédemment8,25. Aucun des stocks de lignées
cellulaires de travail n'a été authentifié ou testé pour les mycoplasmes
récemment, bien que les stocks de semences d'origine utilisés pour créer les
stocks de travail soient exempts de contamination. Les poumons humains pour les
cultures d'AOH ont été acquis en vertu des protocoles approuvés par le Chapel
Hill Institutional Review Board de l'Université de Caroline du Nord. Les
cultures d'AOH représentent un épithélium des voies respiratoires humaines
hautement différencié contenant des cellules épithéliales ciliées et non
ciliées ainsi que des cellules caliciformes. Les cultures sont également
cultivées sur une interface air-liquide pendant plusieurs semaines avant
utilisation, comme décrit précédemment26. En bref, les cellules ont
été lavées avec du PBS et inoculées avec du virus ou simulées diluées dans du
PBS pendant 40 min à 37 ° C. Après l'inoculation, les cellules ont été lavées
trois fois et du milieu frais a été ajouté pour indiquer le temps «0». Trois
réplicats biologiques ou plus ont été récoltés à chaque moment décrit. Aucun
aveuglement n'a été utilisé dans les collections d'échantillons ni les
échantillons ont été randomisés. Toute la culture du virus a été réalisée dans
un laboratoire de niveau de biosécurité (BSL) 3 avec des ventilateurs
redondants dans les armoires de biosécurité, comme décrit précédemment par
notre groupe2. Tout le personnel portait
des respirateurs à adduction d'air filtré (Breathe Easy, 3M) avec des
combinaisons, des tabliers et des bottillons Tyvek et portait un double gant.
Regroupement de séquences et modélisation structurelle.
Les
séquences génomiques complètes et les séquences d'acides aminés des domaines S1
du pic de CoV représentatifs ont été téléchargées à partir de Genbank ou
Pathosystems Resource Integration Center (PATRIC), alignées avec ClustalX et
comparées phylogénétiquement en utilisant l'estimation de la probabilité
maximale à l'aide de 100 bootstrap ou en en utilisant respectivement le package
PhyML (https://code.google.com/p/phyml/) L'arbre a été généré en
utilisant le maximum de vraisemblance avec le package PhyML. La barre d'échelle
représente les substitutions de nucléotides. Seuls les nœuds avec un support
d'amorçage supérieur à 70% sont étiquetés. L'arbre montre que les CoV sont
divisés en trois groupes phylogénétiques distincts définis comme α-CoV, β-CoV et γ-CoV. Les grappes de
sous-groupes classiques sont marquées 2a, 2b, 2c et 2d pour les β-CoV et 1a et 1b pour les α-CoV. Des modèles structurels
ont été générés à l'aide de Modeller (Max Planck Institute Bioinformatics
Toolkit) pour générer des modèles d'homologie pour SHC014 et Rs3367 du SARS RBD
en complexe avec ACE2 sur la base de la structure cristalline 2AJF (Protein Data Bank). Les
modèles d'homologie ont été visualisés et manipulés dans MacPyMol (version
1.3).
Construction de virus
chimériques de type SRAS.
Les
virus de type sauvage et chimérique sont tous deux dérivés du SARS-CoV Urbani
ou du clone infectieux (ic) adapté à la souris (SARS-CoV MA15) comme décrit
précédemment27. Les plasmides contenant des séquences de pointes pour SHC014
ont été extraits par digestion de restriction et ligaturés dans les plasmides E
et F du clone infectieux MA15. Le clone a été conçu et acheté auprès de Bio
Basic sous la forme de six ADNc contigus en utilisant des séquences publiées
flanquées de sites d'endonucléases de restriction de classe II uniques (Bgll).
Par la suite, des plasmides contenant des fragments de génome de type sauvage,
SARS-CoV et SHC014-CoV de type sauvage ont été amplifiés, excisés, ligaturés et
purifiés. Les réactions de transcription in vitro ont ensuite été préformées
pour synthétiser l'ARN génomique complet, qui a été transfecté dans les
cellules Vero E6 comme décrit précédemment2. Le milieu des cellules
transfectées a été récolté et a servi de stock de semences pour les expériences
ultérieures. Les virus chimériques et de pleine longueur ont été confirmés par
analyse de séquence avant utilisation dans ces études. La construction
synthétique du mutant chimérique et du SHC014-CoV pleine longueur a été
approuvée par le comité de biosécurité institutionnelle de l'Université de
Caroline du Nord et le comité de recherche préoccupante à double usage.
Déclaration d'éthique.
Cette
étude a été réalisée conformément aux recommandations pour le soin et
l'utilisation des animaux par l'Office of Laboratory Animal Welfare (OLAW),
NIH. Le Comité institutionnel de protection et d'utilisation des animaux
(IACUC) de l'Université de Caroline du Nord à Chapel Hill (UNC, numéro de
permis A-3410-01) a approuvé le protocole d'étude sur les animaux (IACUC #
13-033) utilisé dans ces études.
Souris et infection in vivo.
Des
souris BALB / cAnNHsD âgées de 10 semaines et de 12 mois ont été commandées
auprès des Laboratoires Harlan. Les infections de souris ont été effectuées
comme décrit précédemment20. En bref, les animaux ont été amenés dans un
laboratoire BSL3 et autorisés à s'acclimater pendant 1 semaine avant
l'infection. Pour l'infection et la vaccination contre le virus vivant atténué,
les souris ont été anesthésiées avec un mélange de kétamine et de xylazine et
infectées par voie intranasale, en cas de provocation, avec 50 μl de solution saline tamponnée au phosphate (PBS) ou virus dilué
avec trois ou quatre souris par point dans le temps, par groupe d'infection par
dose comme décrit dans les légendes des figures. Pour les souris individuelles,
les notations d'infection, notamment le fait de ne pas inhaler la totalité de
la dose, le bouillonnement de l'inoculum par le nez ou l'infection par la
bouche, peuvent avoir conduit à l'exclusion des données de la souris à la
discrétion du chercheur; post-infection, aucun autre critère d'exclusion ou
d'inclusion préétabli n'est défini. Aucun aveuglement n'a été utilisé dans les
expériences animales et les animaux n'ont pas été randomisés. Pour la
vaccination, les souris jeunes et âgées ont été vaccinées par injection de
coussinets plantaires avec un volume de 20 μl de 0,2 μg de vaccin SARS-CoV double-inactivé avec de l'alun ou du faux PBS;
les souris ont ensuite été boostées avec le même régime 22 jours plus tard et
provoquées 21 jours par la suite. Pour tous les groupes, selon le protocole,
les animaux ont été surveillés quotidiennement pour les signes cliniques de la
maladie (courbure, fourrure ébouriffée et activité réduite) pendant la durée de
l'expérience. La perte de poids a été surveillée quotidiennement pendant les 7
premiers jours, après quoi la surveillance du poids s'est poursuivie jusqu'à ce
que les animaux retrouvent leur poids de départ initial ou affichent une prise
de poids continue pendant 3 jours. Toutes les souris qui ont perdu plus de 20%
de leur poids corporel de départ ont été nourries au sol et surveillées
plusieurs fois par jour tant qu'elles étaient sous la limite de 20%. Les souris
qui ont perdu plus de 30% de leur poids corporel de départ ont été
immédiatement sacrifiées selon le protocole. Toute souris jugée moribonde ou
peu susceptible de récupérer a également été sacrifiée sans cruauté à la
discrétion du chercheur. L'euthanasie a été réalisée à l'aide d'une surdose
d'isoflurane et la mort a été confirmée par luxation cervicale. Toutes les
études sur les souris ont été réalisées à l'Université de Caroline du Nord
(assurance du bien-être animal # A3410-01) en utilisant des protocoles
approuvés par le Comité UNC Institutional Animal Care and Use (IACUC).
Analyse histologique.
Le
poumon gauche a été retiré et immergé dans du formol tamponné à 10% (Fisher)
sans gonflement pendant 1 semaine. Les tissus ont été inclus dans de la
paraffine et des coupes de 5 μm ont été préparées par le centre
d'histopathologie UNC Lineberger Comprehensive Cancer Center. Pour déterminer
l'étendue de la coloration de l'antigène, des coupes ont été colorées pour
l'antigène viral en utilisant un anticorps anti-nucléocapside polyclonal
SARS-CoV disponible dans le commerce (Imgenex) et notées de manière aveugle par
pour la coloration des voies respiratoires et du parenchyme comme décrit
précédemment20. Les images ont été capturées à l'aide d'un microscope Olympus
BX41 avec une caméra Olympus DP71.
Essais de
neutralisation de virus.
Des
tests de titre de neutralisation de la réduction de la plaque ont été réalisés
avec des anticorps précédemment caractérisés contre le SRAS-CoV, comme décrit
précédemment11,12,13. En bref, les anticorps
neutralisants ou le sérum ont été dilués en série en deux et incubés avec 100
p.f.u. des différentes souches infectieuses du clone SARS-CoV pendant 1 h à 37
° C. Le virus et les anticorps ont ensuite été ajoutés à une plaque à 6 puits
avec 5 × 105 cellules Vero E6 / puits
avec plusieurs répétitions (n ≥ 2). Après une incubation d'une heure à 37 ° C,
les cellules ont été recouvertes de 3 ml d'agarose à 0,8% dans le milieu. Les
plaques ont été incubées pendant 2 jours à 37 ° C, colorées au rouge neutre
pendant 3 h et les plaques ont été comptées. Le pourcentage de réduction de la
plaque a été calculé comme (1 - (nombre de plaques avec anticorps / nombre de
plaques sans anticorps)) × 100.
Analyse statistique.
Toutes
les expériences ont été menées en contrastant deux groupes expérimentaux (soit
deux virus, soit des cohortes vaccinées et non vaccinées). Par conséquent, des
différences significatives dans le titre viral et la notation histologique ont
été déterminées par un test t de Student bilatéral à des moments individuels.
Les données étaient normalement distribuées dans chaque groupe comparé et
présentaient une variance similaire.
Biosécurité.
Les
études signalées ont été lancées après l'approbation du protocole expérimental
par le Comité institutionnel de biosécurité de l'Université de Caroline du Nord
(Titre du projet: Génération de clones infectieux de CoV de type SARS de
chauve-souris; ID de plan de sécurité de laboratoire: 20145741; annexe G ID:
12279). Ces études ont été lancées avant la pause du financement de la
recherche sur le processus délibératif du gouvernement américain sur certaines
recherches sur le gain de fonction impliquant les virus de la grippe, du MERS
et du SRAS (http://www.phe.gov/s3/dualuse/Documents/gain-of-function.pdf)
Ce
document a été examiné par l'agence de financement, le NIH. La poursuite de ces
études a été demandée, ce qui a été approuvé par le NIH.
SARS-CoV
est un agent sélectif. Tout le travail pour ces études a été effectué avec des
procédures opératoires normalisées (SOP) et des conditions de sécurité
approuvées pour le SRAS-CoV, le MERs-CoV et d'autres CoV associés.
Nos
installations institutionnelles CoV BSL3 ont été conçues pour se conformer aux
exigences de sécurité recommandées par les laboratoires de biosécurité dans les
laboratoires microbiologiques et biomédicaux (BMBL), le département américain
de la Santé et des Services sociaux, le service de santé publique, les Centers
for Disease Control (CDC). ) et le NIH. Des plans de sécurité en laboratoire
ont été soumis au Département de la santé et de la sécurité environnementale
(EHS) et du CDC, et l'installation a été approuvée. Un accès par carte
électronique est requis pour entrer dans l'établissement. Tous les travailleurs
ont été formés par EHS à utiliser en toute sécurité des respirateurs à
adduction d'air filtré (PAPR), et des habitudes de travail appropriées dans un
établissement BSL3 et des plans de surveillance médicale active sont en place.
Nos installations CoV BSL3 contiennent des ventilateurs redondants,
l'alimentation de secours des ventilateurs et des armoires de sécurité
biologique et des congélateurs, et nos installations peuvent accueillir des
racks de souris SealSafe. Les matières classées comme agents BSL3 sont
constituées de souches de précurseurs du SRAS-CoV, de bat CoV, de MERS-CoV et
de mutants dérivés de ces agents pathogènes. Dans les installations BSL3,
l'expérimentation de virus infectieux est effectuée dans une armoire de
biosécurité certifiée de classe II (BSC). Tous les membres du personnel portent
des gommages, des costumes et des tabliers Tyvek, des PAPR et des
couvre-chaussures, et leurs mains sont à double gant. Les utilisateurs de BSL3
sont soumis à un plan de surveillance médicale surveillé par la Clinique de
santé au travail des employés de l'Université (UEOHC), qui comprend une
vaccination annuelle contre la grippe physique et annuelle et la déclaration
obligatoire de tout symptôme associé à l'infection à CoV pendant les périodes
de travail dans le BSL3. Tous les utilisateurs de BSL3 sont formés à la gestion
de l'exposition et aux protocoles de déclaration, sont prêts à s'auto-mettre en
quarantaine et ont été formés pour une livraison en toute sécurité à un service
local de gestion des maladies infectieuses en cas d'urgence. Tous les
événements d'exposition potentiels sont signalés et examinés par l'EHS et
l'UEOHC, avec des rapports déposés à la fois au CDC et au NIH.
Traduction automatique
-------------------------------
NOTES de H. G.
Hannibal GENSÉRIC
 Figure 1: Les virus de
type SRAS se répliquent dans les cellules des voies respiratoires humaines et
produisent une pathogenèse in vivo.
Figure 1: Les virus de
type SRAS se répliquent dans les cellules des voies respiratoires humaines et
produisent une pathogenèse in vivo.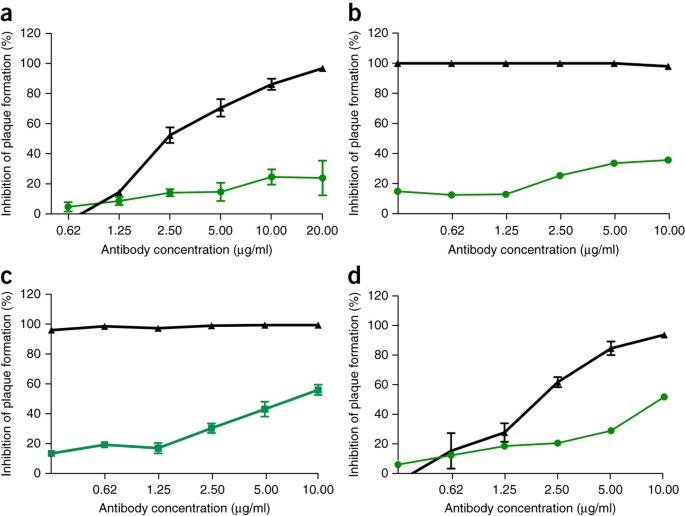


des preuves que les usa sont des criminels ...vietnam,Cambodge, laos , Lybie, Irak, kossovo, Ukraine Syrie, liban, palestine etc..etc....
RépondreSupprimerVraiment le gouvernement américain est sans morale et sans coeur
RépondreSupprimer